Laboratoř regulačních RNA
Vedoucí:
Mgr. Jarmila Hnilicová, Ph.D.
Kde nás najdete:
Budova Viničná 5, Praha 2 - Nové Město, 128 43
přízemí (místnost 006)
Výzkumná problematika
Další informace lze nalézt i na adrese https://web.natur.cuni.cz/molbio/hnilicovalab/
Laboratoř regulačních RNA vznikla jako nejmladší výzkumná skupina katedry genetiky a mikrobiologie na konci roku 2023. Předtím byla součástí Laboratoře mikrobiální genetiky a genové exprese na Mikrobiologickém ústavu AV ČR, v.v.i. Skupina se zabývá malými nekódujícími RNA, které regulují bakteriální transkripci. Zajímají nás hlavně RNA, které se váží na RNA polymerázy (RNAP). Naším dlouhodobým cílem je pochopit, jak tyto RNA fungují, a zjistit, zda by mohly být v budoucnu využity pro vývoj specifických RNA antibiotik zaměřených na jednotlivé druhy bakterií.
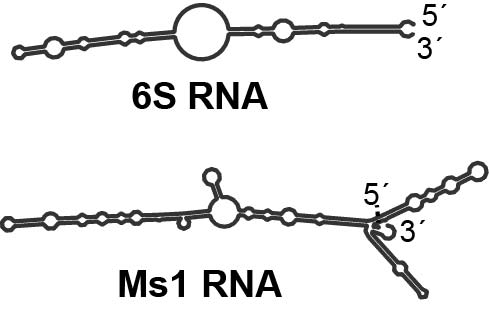 Již v r. 2013 jsme objevili, že Ms1 RNA, což je RNA přítomná ve značném množství během stacionární fáze životního cyklu bakterie Mycobacterium smegmatis, se dokáže vázat k proteinům core RNAP bez pomoci sigma faktorů, což byl teprve druhý příklad RNA, která je toho schopna (po 6SRNA, která se v M. smegmatis nevyskytuje). Navíc jsme ukázali, že Ms1 RNA reguluje množství RNAP u této bakterie.
Již v r. 2013 jsme objevili, že Ms1 RNA, což je RNA přítomná ve značném množství během stacionární fáze životního cyklu bakterie Mycobacterium smegmatis, se dokáže vázat k proteinům core RNAP bez pomoci sigma faktorů, což byl teprve druhý příklad RNA, která je toho schopna (po 6SRNA, která se v M. smegmatis nevyskytuje). Navíc jsme ukázali, že Ms1 RNA reguluje množství RNAP u této bakterie.
Laboratoř používá k detekci RNA interagujících s RNAP u různých bakteriích metodu nativního RIP-seq. Ve spolupráci s bioinformatiky dokážeme rozlišit nově vznikající transkripty asociované s transkribujícími RNAP od RNA které se važí na netranskribující RNAP. Ve spolupráci s Ministerstvem obrany ČR jsme provedli (v jejich BSL3 laboratoři) RIP-seq i sM. tuberculosis a ukázali jsme, že homolog Ms1, MTS2823, i v tomto případě interaguje s RNAP během stacionární fáze životního cyklu této bakterie. M. smegmatis RIP-seq aM. smegmatis NGS data jsou nyní veřejně dostupná na adrese http://msmegseq.elixir-czech.cz/.
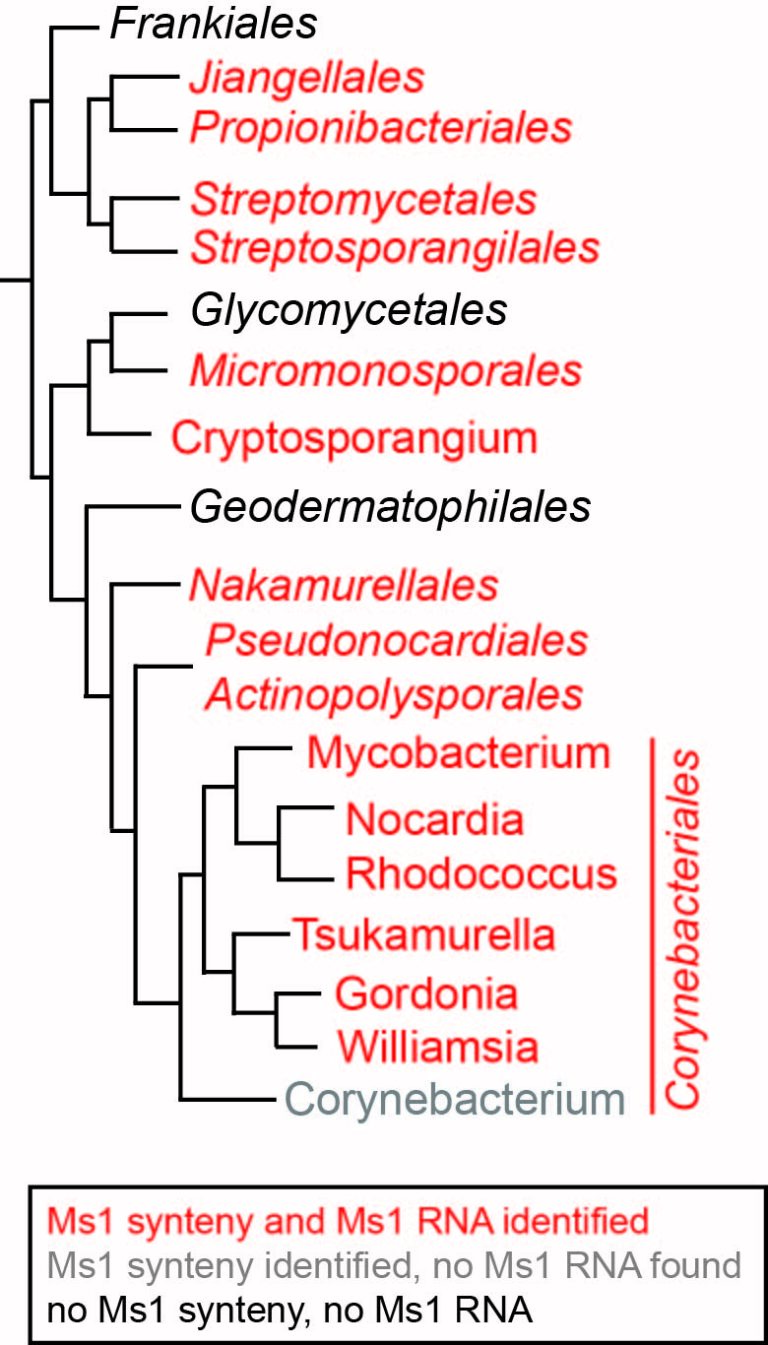 Další otázkou bylo, jestli je Ms1 RNA přítomna i u jiných bakterií než pouze Mycobacterium sp. Opět s pomocí bioinformatických přístupů se podařilo identifikovat homology Ms1 u řady aktinobakterií a u Streptomyces coelicolor. Překvapivě však jsme u několika druhů bakterií (např. Corynebacterium glutamicum) nedetekovali přítomnost ani 6S RNA, ani Ms1 RNA. Nicméně pomocí RIP-seq se nám u C. glutamicum podařilo objevit novou, cca 500 nukleotidů dlouhou RNA se schopností vázat se na obě formy RNAP: RNAP core a RNAP holoenzym (s primárním sigma faktorem). Tato RNA má unikátní sekundární strukturu a délku, nepodobnou 6S i Ms1 RNA, takže se nedá detekovat pouze bioinformatickými přístupy. Je možné, že další bakterie, které neobsahují 6S/Ms1/CoRP RNA, mohou mít další nové typy RNAP-vazebných RNA. Identifikace těchto dalších RNA přispěje k našemu porozumění variabilitě RNA struktur schopných asociovat s RNAP.
Další otázkou bylo, jestli je Ms1 RNA přítomna i u jiných bakterií než pouze Mycobacterium sp. Opět s pomocí bioinformatických přístupů se podařilo identifikovat homology Ms1 u řady aktinobakterií a u Streptomyces coelicolor. Překvapivě však jsme u několika druhů bakterií (např. Corynebacterium glutamicum) nedetekovali přítomnost ani 6S RNA, ani Ms1 RNA. Nicméně pomocí RIP-seq se nám u C. glutamicum podařilo objevit novou, cca 500 nukleotidů dlouhou RNA se schopností vázat se na obě formy RNAP: RNAP core a RNAP holoenzym (s primárním sigma faktorem). Tato RNA má unikátní sekundární strukturu a délku, nepodobnou 6S i Ms1 RNA, takže se nedá detekovat pouze bioinformatickými přístupy. Je možné, že další bakterie, které neobsahují 6S/Ms1/CoRP RNA, mohou mít další nové typy RNAP-vazebných RNA. Identifikace těchto dalších RNA přispěje k našemu porozumění variabilitě RNA struktur schopných asociovat s RNAP.





















